Narzędzie Tiara – nazwa inspirowana tiarą przydziału z Harrego Pottera. Przeczytajcie co mówią o tym sami autorzy
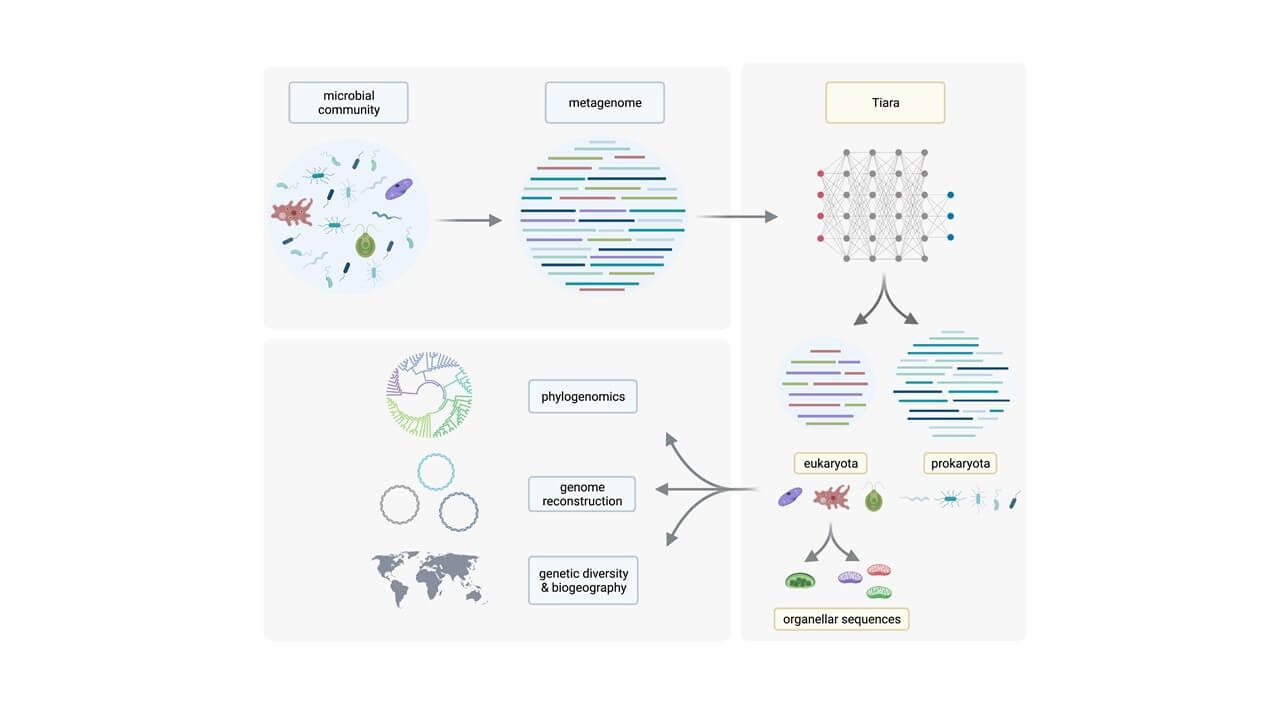
Naukowcy z Instytutu Biologii Ewolucyjnej są autorami nowego narzędzia bioinformatycznego do klasyfikacji sekwencji z danych metagenomowych. “Inspiracją do nadania narzędziu nazwę Tiara była słynna tiara z Harrego Pottera, za pomocą której uczniowie bajkowej szkoły Hogwart byli przydzielani na podstawie swoich naturalnych predyspozycji do domów w Szkole Magii i Czarodziejstwa. Stworzony przez nas program umożliwia rozdział sekwencji na: eukariotyczne (w tym organellarne) i prokariotyczne” – mówią twórcy programu.
Doktorant mgr Michał Karlicki i student Stanisław Antonowicz wraz z dr hab. Anną Karnkowską z Instytutu Biologii Ewolucyjnej Wydziału Biologii UW są autorami artykułu naukowego, w którym omawiają nowo opracowane narzędzie oparte o metody uczenia maszynowego do szybkiej i dokładnej klasyfikacji sekwencji eukariotycznych i prokariotycznych. Publikacja ukazała się na łamach czasopisma Bioinformatics.
Artykuł „Tiara: deep learning-based classification system for eukaryotic sequences” powstał w ramach badań finansowanych z EMBO Installation Grant kierowanego przez dr hab. Annę Karnkowską.
Wraz z rosnącą liczbą danych metagenomowych pochodzących z sekwencjonowania wysokoprzepustowego poznajemy coraz lepiej różnorodność i funkcje mikroorganizmów wielu środowisk. Dużym wyzwaniem jest jednak odzyskiwanie i prawidłowa klasyfikacja sekwencji eukariotycznych. Naszym celem było przygotowanie narzędzia, które dokładnie i szybko identyfikuje sekwencje eukariotyczne wraz z podziałem na sekwencje pochodzące z genomu jądrowego, mitochondrialnego i chloroplastowego, co jest niezbędnym krokiem w kierunku lepszego zrozumienia różnorodności eukariota.
Opracowane przez nas narzędzie o nazwie Tiara, opiera się na uczeniu maszynowym i służy przede wszystkim do identyfikacji sekwencji eukariotycznych w danych metagenomowych. Zastosowaliśmy w nim dwuetapowy proces klasyfikacji, co umożliwia identyfikację sekwencji jądrowych i organellarnych, a następnie dzieli sekwencje organellarne na plastydowe i mitochondrialne. Przeprowadzone testy wykazały, że Tiara dokładniej klasyfikuje sekwencje eukariotyczne niż inne dostępne narzędzia i jest jedynym programem, który specyficznie identyfikuje sekwencje organellarne. Tiara dobrze radzi sobie zarówno z danymi pochodzącymi z mikrobiomów pojedynczych komórek eukariotycznych, jak i z dużymi i złożonymi zestawami danych obejmującymi różnorodność mikroorganizmów w oceanach. Narzędzie i wskazówki dotyczące instalacji oraz użytkowania znajdują się w ogólnodostępnym repozytorium GitHub
https://ibe-uw.github.io/tiara/.
Zachęcamy do korzystania, a autorom serdecznie gratulujemy! Chciałoby się powiedzieć: no to “tiarujemy”! 😉