Zespoły mikroorganizmów słodkowodnych w gradiencie eutrofizacji: różnorodność i interakcje protistów i bakterii (MicroDivEr)

Mikroorganizmy są niezwykle liczną i różnorodną grupą, obecną we wszystkich ekosystemach. Zaliczamy do nich organizmy bezjądrowe (prokariotyczne), takie jak bakterie i archeony, oraz jednokomórkowe organizmy jądrowe (eukariotyczne), zwane też protistami. Zespoły mikroorganizmów stanowią kluczowy element ekosystemów, a ich funkcjonowanie zależy nie tylko od składu taksonomicznego zespołu, ale również od występujących w nim interakcji.
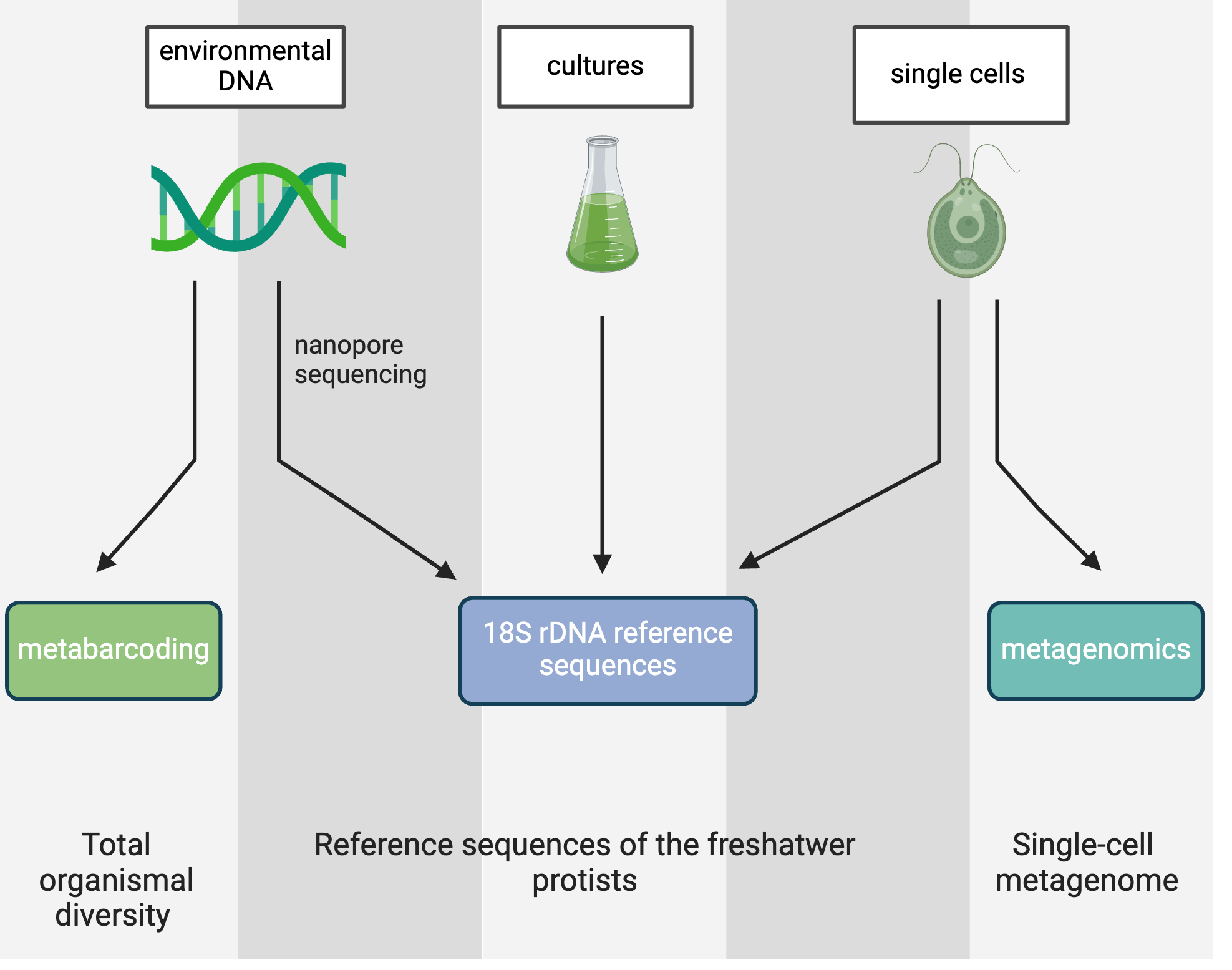
W realizowanym projekcie identyfikujemy mikroorganizmy eukariotyczne i bakterie występujące w jeziorach Pojezierza Mazurskiego. Wykorzystujemy do tego metody sekwencjonowania krótkich i fragmentów DNA. Żeby prawidłowo zidentyfikować organizmy na podstawie sekwencji DNA potrzebna jest baza referencyjna. Poszerzymy istniejącą bazę stosując sekwencjonowanie pojedynczych komórek i sekwencjonowanie oparte na nanoporach. Analiza współwystępowania protistów i bakterii umożliwi identyfikację ich interakcji. Poznanie sekwencji genomów protistów i towarzyszących im bakterii pozwoli zaś na wgląd w naturę tych interakcji. Szczególnie interesują nas relacje protistów i endosymbiotycznych bakterii oraz bakterii patogennych. Na koniec określimy jak czynniki środowiskowe wpływają na zróżnicowanie zespołów mikroorganizmów i ich interakcje.
Przeprowadzone badania przybliżą nas do poznania różnorodności i interakcji mikroorganizmów w wodach słodkich. Identyfikacja interakcji protistów i patogennych bakterii pomoże określić naturalny rezerwuar tych bakterii. Ułatwi to przewidywanie ich niebezpiecznych dla zdrowia człowieka i zwierząt pojawów.Badania w jeziorach o różnym statusie troficznym umożliwią natomiast ocenę wpływu użyźnienia jezior na skład zespołów mikroorganizmów. Pomoże to przewidzieć zmiany w ekosystemach słodkowodnych wynikające z działalności człowieka i przyczyni się do ich skuteczniejszej ochrony.
Pobór prób i analiza różnorodności zespołów mikroorganizmów
W latach 2021-2024 pobieraliśmy próby z kilkunastu jezior Pojezierza Mazurskiego różniących się od siebie statusem troficznym i poziomem antropopresji. Dla wszystkich jezior pobieraliśmy próby trzy razy do roku, tak aby uwzględnić zmiany sezonowe zachodzące w jeziorach strefy umiarkowanej. Zebrany materiał posłużył nam do analiz własności fizyko-chemicznych wody oraz wyizolowania całościowego DNA mikroorganizmów jeziornych. Zebraliśmy również materiał do badań na pojedynczych komórkach, który umożliwi nam wgląd w badanie interakcji między protistami i bakteriami na poziomie pojedynczych komórek.


Badania różnorodności mikroorganizmów w jeziorach Pojezierza Mazurskiego
Badania interakcji mikroorganizmów
Publikacje
- Małgorzata Chwalińska, Michał Karlicki, Sarah Romac, Fabrice Not, Anna Karnkowska. From short to long reads: enhanced protist diversity profiling via Nanopore metabarcoding. Metabarcoding and Metagenomics (MBMG), Volume 9 , October 2025, e163750. doi:https://doi.org/10.3897/mbmg.9.163750
- Metody Hollender, Marta Sałek, Michał Karlicki, Anna Karnkowska. Single-cell genomics revealed Candidatus Grellia alia sp. nov. as an endosymbiont of Eutreptiella sp. (Euglenophyceae). Protist, Volume 175, Issue 2, April 2024, 126018. https://doi.org/10.1016/j.protis.2024.126018
- Michał Karlicki, Anna Bednarska, Paweł Hałakuc, Kacper Maciszewski, Anna Karnkowska. Spatio-temporal changes of small protist and free-living bacterial communities in a temperate dimictic lake: insights from metabarcoding and machine learning. FEMS Microbiology Ecology, Volume 100, Issue 8, August 2024, fiae104. https://doi.org/10.1093/femsec/fiae104

- Badania zespołów protistów i bakterii w jeziorze Roś wykazały wyraźne wzorce sezonowe.
- Dzięki analizie danych z wykorzystaniem uczenia maszynowego, zidentyfikowano organizmy, które mimo że nie dominują to kształtują zespół mikroorganizmów charakterystyczny dla danego sezonu.
- Oddzielny zespół stanowiły organizmy charakterystyczne dla głębokich, beztlenowych warstw jeziora w czasie letniej stratyfikacji.
- Poznanie wzorców sezonowych zmian mikroorganizmów pozwala lepiej przewidzieć skutki wydłużonego okresu letniej stratyfikacji, który obserwujemy wraz z ociepleniem klimatu.
- Opracowaliśmy ścieżkę analizy danych, która umożliwia dokładny opis różnorodności protistów i bakterii na podstawie danych amplikonowych
Dane i programy wytworzone w ramach realizacji projektu
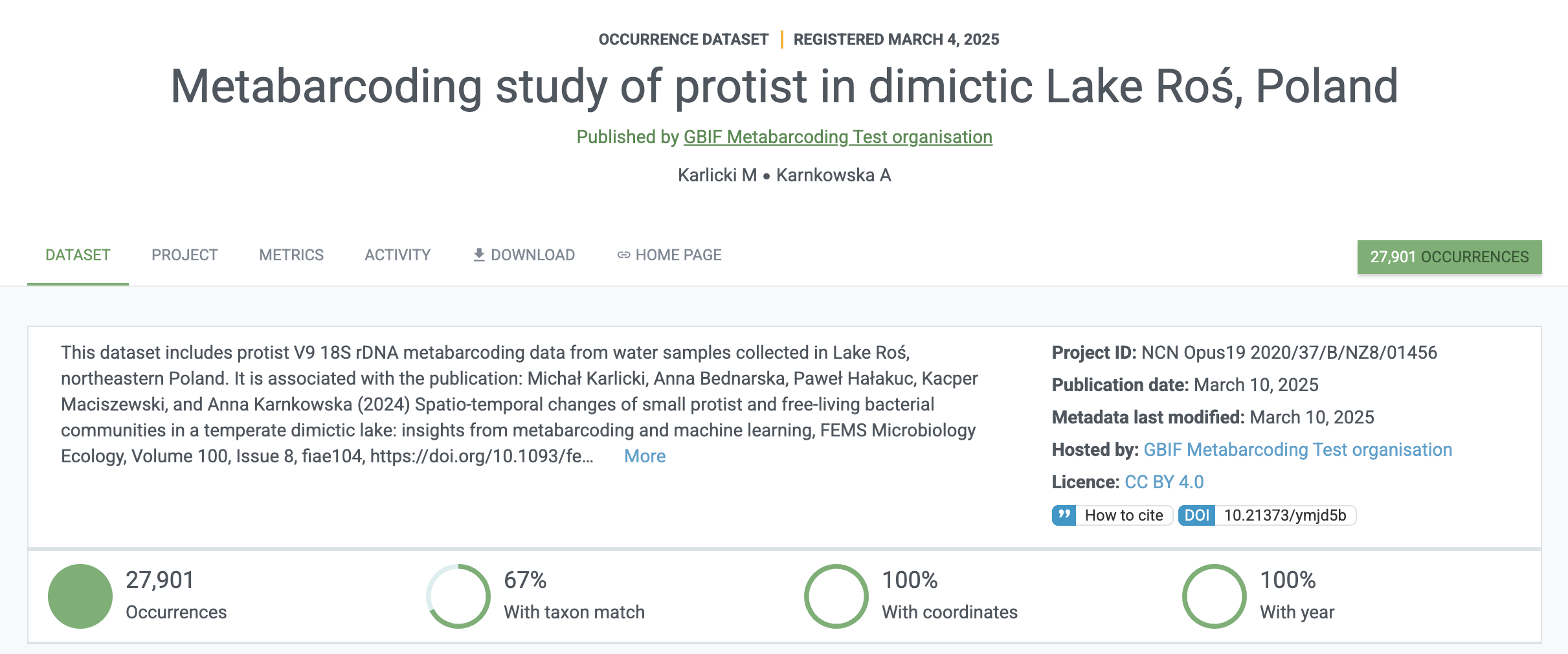
Dane z badań przeprowadzonych na jeziorze Roś są już dostępne w publicznych bazach danych! Bardzo cieszymy się, że nasze dane są też dostępne w bazie GBIF w ramach pilotażowego etapu poszerzania tej bazy o dane molekularne. Dane znajdują się pod tym adresem: https://www.gbif-uat.org/dataset/ce94b68c-abb0-4d75-abe9-2bfc88431906
Wszystkie otwarte dane poza dedykowanymi repozytoriami będą dostępne w interdyscyplinarnym repozytorium przechowywania danych Zenodo. Projekt MicroDivEr masz już swoją stronę, która będzie aktualizowana wraz z publikowaniem kolejnych danych.
Doniesienia konferencyjne
- Sałek M., Hollender M., Hałakuc P., Karlicki M., Maciszewski K., Karnkowska A. Finding interaction patterns between protists and prokaryotes based on a single cell microbiome analysis. 51st Jírovec’s Protozoological Days, Svratka, Czechy; 20-24.06.2022 – plakat
- Chwalińska M., Karlicki M., Sałek M., Smacchia V., Hałaku P., Maciszewski K., Karnkowska A. Freshwater protists diversity elucidated with the short Illumina vs long MinION amplicons of 18S rRNA gene. 51st Jírovec’s Protozoological Days. Hotel Svratka, Czechy; 20-24.06.2022 – wystąpienie ustne
- Karlicki M., Chwalińska M., Bednarska A., Hałakuc P., Maciszewski K., Karnkowska. Spatiotemporal dynamics of eukaryotic communities in a model dimictic lake. 51st Jírovec’s Protozoological Days. Hotel Svratka, Czechy; 20-24.06.2022 – wystąpienie ustne
- Hollender M., Sałek M., Hess S., Karnkowska A. “Metagenomic and fluorescence in situ hybridization analyses of Eutreptiella endosymbiont. 2nd Annual International Congress on Euglenoids; 7-10.11.2022 – wystąpienie ustne
- Sałek M., Hollender M., Hałakuc P., Karlicki M., Maciszewski K., Karnkowska A. Finding interaction patterns between protists and eukaryotes based on single cell microbiome approach: obtaining good quality data for computational analyses. Meeting of the EMBO Young Investigator Network on computational methods in ecology and evolutionary biology of microbes, Chęciny, Poland; 7-9.07.2022 – wystąpienie ustne
- Hollender M., Sałek M., Karlicki M., Karnkowska A. Workflow for genome recovery of protistan prokaryotic symbionts from single-cell data. Meeting of the EMBO Young Investigator Network on computational methods in ecology and evolutionary biology of microbes, Chęciny, Poland; 7-9.07.2022 – wystąpienie ustne
- Hollender M., Sałek M., Karlicki M., Karnkowska A. Endosymbiont for each protist – protocol for prokaryotic endosymbionts identification from protists’ single cells. ISEP Virtual Meeting. Online; 19-20.01.2023 – poster
- Chwalińska M., Karlicki M., Karnkowska A. Freshwater protist diversity analysis using long nanopore 18S rDNA amplicons reveals hidden diversity of ‘Excavates’. ECOP-ISOP joint meeting “The Century of Protists”. Vienna, Austria; 9-14.07.2023 – poster
- Hollender M., Karnkowska A. A new group of endosymbiotic Legionellales of Euglenophyceae identified via metagenomic analyses. ECOP-ISOP joint meeting “The Century of Protists”. Vienna, Austria; 9-14.07.2023 – poster
- Sałek M., Chwalińska M., Hollender M., Smacchia V., , Karnkowska A. Abundance and diversity of protistan-prokaryotic interactions in freshwater lakes of the Masurian Lakeland. Symposium on Aquatic Microbial Ecology – SAME17. Tartu, Estonia; 20-25.08.2023 – poster
- Hollender M., Sałek M., Karnkowska A. Protist single cells picked from environmental samples allow to uncover new bacterial endosymbionts. Meeting of the EMBO Young Investigator Network on computational methods in ecology and evolutionary biology of microbes, Chęciny, Poland; 5-7.10.2023 – wystąpienie ustne
- Sałek M., Chwalińska M., Hollender M., Smacchia V., , Karnkowska A. Abundance and diversity of protistan-prokaryotic interactions in freshwater lakes of the Masurian Lakeland based on single cell microbiome study. Meeting of the EMBO Young Investigator Network on computational methods in ecology and evolutionary biology of microbes, Chęciny, Poland; 5-7.10.2023 – wystąpienie ustne
- Sałek M., Hollender M., Smacchia V., Karnkowska A. Exploring protist-bacteria interactions along eutrophication gradient: insights from amplicon and single-cell analyses in freshwater ecosystems. 2024 PSA-ISOP-ISEP Joint Meeting, Seattle, USA, 29.07-1.08.2024 – wystąpienie ustne
- Karnkowska, A. Exploring protist endosymbioses: Insights into diversity, ecology, and evolution. Haltern am See, Niemcy. 27.02-1.03 2024, Zaproszony wykład.
- Karnkowska, A. Let’s get together and feel all right: Bacterial endosymbioses abundance in protists. Comparative genomics of unicellular eukaryotes: Interactions and symbioses. Sant Feliu de Guixols, Hiszpania. 30.09-5.10 2024. Zaproszony wykład.
- Karlicki, M.,Woodhouse, J., Grossart, HP., Piwosz, K., Karnkowska., A. Plastid-genome resolved metagenomics reveals diversity and ecology of eukaryotic phytoplankton in the long-term study of freshwater lakes, Kapsztad, RPA, 18.08 – 23.08. 2024. Prezentacja ustna
- Hollender M., Sałek M., Karnkowska A. Overlooked diversity of bacterial endosymbionts explored through single-cell genomics of freshwater protists. 53rd Jírovec’s Protozoological Days, Karlova Studanka, Czechy, 29.04-03.05.2024. Prezentacja ustna.
- Smacchia V, Karlicki M, Sałek M, Chwalińska M, Garcia Cunchillos I, Karnkowska A. Diversity of microbial communities along the eutrophication gradient in the Masurian lakes, northeastern Poland. IUMS2024: Microorganisms for sustainable solutions: environmental & clinical implementations. Florencja, Wlochy, 23.10.-25.10.2024. Poster.
Prace dyplomowe
Metody Hollender (2024) Genomika pojedynczych komórek jako narzędzie do badań prokariotycznych endosymbiontów Protista. Praca magisterska
Małgorzata Chwalińska (2022) Porównanie krótkich i długich amplikonów genu 18S rDNA w analizie różnorodności słodkowodnych protistów. Praca magisterska
Upowszechnianie wyników projektu