Ewolucja i funkcja odwróconych powtórzeń (IR) w genomach plastydowych Euglenophyta
Plastydy to organella komórkowe obecne u roślin, ale także u licznych, często daleko spokrewnionych ze sobą grup glonów. Organella te powstały na drodze endosymbiozy z cyjanobakteriami i umożliwiły komórkom eukariotycznym prowadzenie procesu fotosyntezy. Plastydy posiadają własny materiał genetyczny, który zazwyczaj uorganizowany jest w pojedynczą, kolistą cząsteczkę DNA. Pomimo ich szerokiego rozprzestrzenienia na znacznej części drzewa życia i, w konsekwencji, wśród rozmaitych gospodarzy o różnej historii ewolucyjnej, większość genomów plastydowych charakteryzuje się względnie wysokim stopniem konserwacji zawartości genów oraz struktury. Podczas gdy geny kodujące białka zazwyczaj występują w genomach plastydowych w pojedynczych kopiach, operon rybosomalny, złożony z dwóch genów kodujących małą (16S) i dużą (23S) podjednostkę rybosomu, przeważnie występuje w dwóch kopiach o przeciwnej orientacji. Fragmenty genomu plastydowego zawierające ten operon, a także dwa lub więcej genów tRNA, a w niektórych przypadkach również geny kodujące białka, nazywane są odwróconymi powtórzeniami (ang. inverted repeats, IR). Powtórzenia te dzielą pozostałą część genomu plastydowego na mały (ang. small single-copy SSC) oraz duży region jednokopijny (ang. large single-copy, LSC), tworząc układ nazywany strukturą czterodzielną.
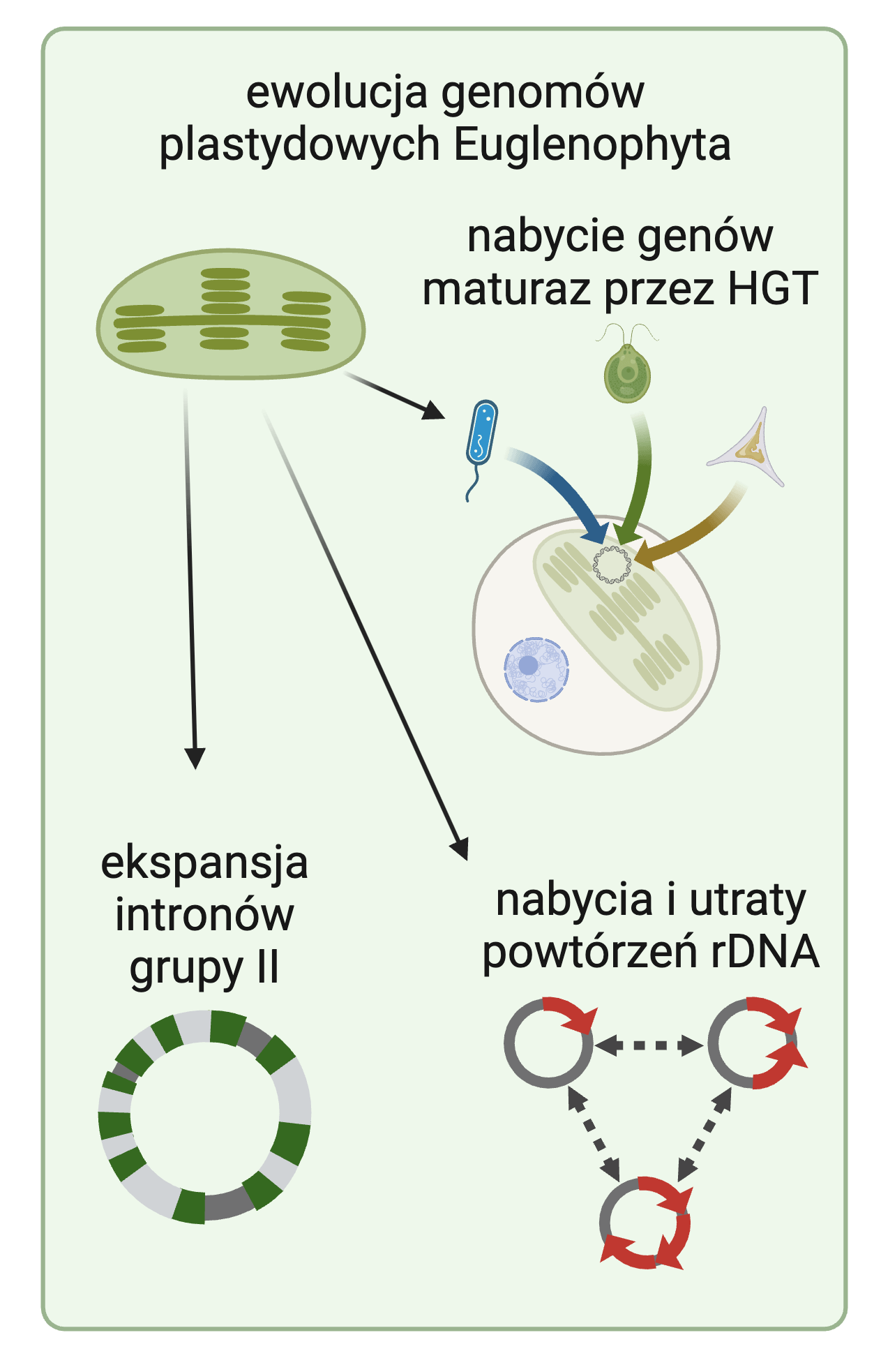
Pomimo rosnącej dostępności technologii sekwencjonowania genomów oraz wzrostu zainteresowania dziedziną genomiki, wiele problemów dotyczących genomów plastydowych, ich działania oraz ewolucji, wciąż pozostaje nierozwiązanych. Wśród głównych wyzwań w badaniach genomów plastydowych znajdują się wyjaśnienie utrzymywania czterodzielnej struktury genomu w przeważającej większości plastydów oraz charakterystyka zawartości pierwotnych genomów plastydowych dla grup posiadających plastydy. Do pierwszego z powyższych problemów istnieją odniesienia w najnowszej literaturze: aktualna hipoteza głosi, że odwrócone powtórzenia pełnią rolę w naprawie mutacji, co wspiera obserwacja, że tempo mutacji jest niższe w genach zlokalizowanych w IR niż w genach jednokopijnych. Dotychczas nie zbadano jednak, czy tę samą regularność można zaobserwować w nietypowych wariantach powtórzeń zawierających geny rybosomalne, takich jak powtórzenia tandemowe, gdzie operon rybosomalny obecny jest w następujących bezpośrednio po sobie kopiach o jednakowej orientacji.
Celem projektu było pogłębienie stanu wiedzy na temat ewolucji materiału genetycznego chloroplastów (ptDNA) u jednokomórkowych glonów – euglenin. Uzyskane dane obejmowały kompletne genomy chloroplastowe dwunastu szczepów euglenin, zarówno morskich, jak i słodkowodnych, reprezentujących niemal cały zakres różnorodności biologicznej wybranej grupy. Na podstawie powyższych danych udało się wyciągnąć szereg wniosków dotyczących ewolucji badanych organizmów, jak również ewolucji genomów plastydowych jako takich. Analizy ewolucji struktury ptDNA euglenin pozwoliły zaobserwować, że grupa ta cechuje się niezwykle dynamicznie zmienną organizacją materiału genetycznego chloroplastów. Oprócz konwencjonalnej struktury czterodzielnej, opisanej m.in. w genomach chloroplastowych roślin, wśród euglenin występują również dwa inne sposoby organizacji ptDNA. Wzór rozprzestrzenienia nietypowych struktur genomów chloroplastowych wśród dalece spokrewnionych gatunków euglenin sugeruje, że przemiany tych struktur zachodziły w toku ewolucji wielokrotnie na drodze nabywania, tracenia, a także zmiany orientacji całych grup genów. Wniosek ten pozwala poddać w wątpliwość, czy teza o silnej konserwacji ewolucyjnej struktury czterodzielnej, wysunięta w oparciu o badania genomów chloroplastowych roślin, znajduje poparcie wśród innych grup organizmów posiadających chloroplasty. Co ciekawe, w genomach chloroplastowych euglenin zidentyfikowano nieznaną wcześniej różnorodność enigmatycznej rodziny genów kodujących maturazy intronów grupy II. Produktami tych genów są białka umożliwiające wycinanie sekwencji niekodujących z genów, ale także ich retrotranspozycję, czyli wbudowanie tejże sekwencji niekodującej w inne miejsce w genomie, na przykład w obręb innego genu. Geny maturaz okazały się być nie tylko szerzej rozprzestrzenione wśród euglenin, niż sądzone wcześniej, ale także liczniejsze – opisano bowiem dwa nowe dla nauki geny (mat6 oraz mat7), jak dotąd zidentyfikowane wyłącznie w pozornie zredukowanych genomach chloroplastowych euglenin. Ponadto, zaobserwowano, że geny kodujące maturazy mogą charakteryzować się unikatową organizacją przestrzenną, polegającą na obecności genu maturazy wewnątrz intronu w innym genie maturazy, który z kolei kodowany jest wewnątrz intronu w jeszcze innym genie. Struktura ta, nazwaną „maturioszką” – matrioszką z maturaz, również nie została wcześniej zaobserwowana u żadnego znanego organizmu.
Powyższe wnioski stanowią istotne dopełnienie aktualnego stanu wiedzy na temat ewolucji materiału genetycznego organelli, takich jak chloroplasty. Pozwalają one sądzić, że mimo niewielkiego rozmiaru i stosunkowo nielicznych biologicznych funkcji, przemiany ewolucyjne ptDNA mogą być niezwykle złożone oraz bogate w nieznane dotychczas procesy. Co więcej, umacniają one znaną, jednak często zaniedbywaną tezę, że organizmy modelowe w badaniach nad ewolucją biologiczną nie zawsze wystarczają do zrozumienia zjawisk na poziomie globalnym, a wszelkie modele biologiczne wymagają ciągłej rewizji oraz usprawniania, aby przybliżyć nas do zrozumienia ewolucji życia na Ziemi.
Uzyskane wyniki zostały przedstawione w dwóch publikacjach naukowych:
- Maciszewski K, Dabbagh N, Preisfeld A, Karnkowska A (2022a). Maturyoshka: A maturase inside a maturase, and other peculiarities of the novel chloroplast genomes of marine euglenophytes. Molecular Phylogenetics and Evolution, 170: 107441.
- Maciszewski K, Fells A, Karnkowska A (2022b) Challenging the importance of plastid genome structure conservation: new insights from euglenophytes. Molecular Biology and Evolution, 39(12): msac255. DOI: 10.1093/molbev/msac255.